Биоинформатика: метод во главе угла
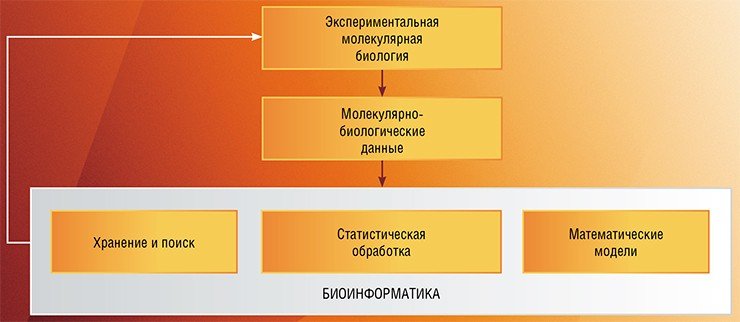
Информационные технологии начали использоваться для хранения и анализа биологических данных (прежде всего данных молекулярной биологии) уже с 60-х гг. прошлого века. Это было вызвано быстрым накоплением информации о строении сложных органических полимеров – нуклеиновых кислот, обеспечивающих хранение и передачу наследственной информации, и белков, играющих в клетке структурную, ферментативную и регуляторную роль.
Но бурное развитие биоинформатика получила лишь спустя три десятилетия, на волне появления мощной вычислительной техники и сетевых коммуникативных технологий, обеспечивших хранение огромных массивов молекулярно-биологических данных и свободный доступ к ним для любого исследователя
Основателем биоинформатики как нового научного направления можно считать американскую исследовательницу М. Дайхофф, которая собрала в своем «Атласе белковых последовательностей и структур» (1965) первые данные об аминокислотных последовательностях этих макромолекул. Их анализ позволили Дайхофф сформулировать математическую модель эволюции белков, на основе которой и осуществлялся в дальнейшем поиск родственных последовательностей и их классификация.
Таким образом была заложена основная парадигма биоинформатики: разработка инструментов компьютерного представления биологических данных, обеспечение их хранения и доступности; статистическая обработка результатов экспериментов и реконструкция на этой основе математических и компьютерных моделей биологических процессов. Это определило место нового направления среди других биологических дисциплин: с помощью биоинформационного подхода появилась возможность уточнять существующие модели биологических систем и создавать новые, на основе которых можно планировать эксперименты.
Появление в 1990-х гг. технологий секвенирования позволило ученым «читать» нуклеотидные последовательности геномов организмов, от простых (вирусы и бактерии) до сложноорганизованных (животных и растений). Наконец, в 2001 г. был секвенирован первый геном человека – это событие ознаменовало собой начало новой эпохи так называемых постгеномных исследований, когда основной задачей биологии стала интерпретация геномных данных, в том числе распознавание функций генов и их роли в живых организмах.
К настоящему времени секвенировано уже более 4,3 тыс. геномов, в том числе около 180 геномов высших организмов (эукариот), а секвенирование еще нескольких тысяч геномов будет завершено в ближайшее время. И в анализе всех этих данных основная роль отводится биоинформатике, в которой сегодня можно выделить несколько основных направлений:
• анализ данных высокопроизводительных экспериментов по секвенированию геномов;
• анализ геномных последовательностей (аннотация генома, предсказание сайтов связывания ДНК, предсказание функций генов);
• структурная биоинформатика;
• компьютерная системная биология (генные сети, регуляторные сети);
• эволюционная биоинформатика;
• компьютерный анализ текстов.
Нуклеотидный пазл
Как известно, геном человека представляет собой набор молекул ДНК общим размером 3×109 нуклеотидов, а длина отдельных хромосом варьирует от 50 до 250 млн нуклеотидов. В процессе высокопроизводительного секвенирования генома молекулы ДНК дробятся на короткие (50—200 нуклеотидов) фрагменты ДНК, последовательность которых можно автоматически идентифицировать.
В результате получаются большие массивы данных, представляющие собой результат расшифровки коротких последовательностей во множестве копий, полностью или частично перекрывающихся между собой. Для того чтобы реконструировать весь геном, нужно решить обратную задачу – собрать из этих фрагментов полные нуклеотидные последовательности, составляющие отдельные хромосомы.
Для решения задачи ассемблирования (сборки) генома имеется два принципиальных подхода. Во-первых, сборку последовательностей можно вести «вслепую», на основании лишь известных фрагментов (метод сборки de novo). В этом случае используется тот факт, что благодаря перекрыванию коротких фрагментов одна и та же последовательность ДНК может быть «покрыта» многократно.
Такой подход оправдан в случае, если геном организма неизвестен. Основной проблемой при этом является наличие в геноме большого числа одинаковых последовательностей, определить точное местоположение которых методами одной лишь биоинформатики невозможно. Бактерии содержат мало таких повторяющихся участков, поэтому сборка их геномов по данным секвенирования осуществляется с высокой (до 99 %) точностью. Однако для высших организмов характерен избыток повторенной ДНК, что существенно затрудняет сборку геномов de novo из коротких фрагментов. В результате приходится применять более трудоемкие и дорогие экспериментальные методы, позволяющие получить фрагменты большей (до тысячи нуклеотидов) длины.
Другой подход используется тогда, когда геном вида, к которому принадлежит организм, уже секвенирован. В этом случае требуется только определить положение отдельных секвенированных фрагментов в известной последовательности. Такая процедура «картирования» намного проще, чем сборка de novo, однако и она требует применения специальных алгоритмов из-за огромного размера данных (типичная задача – картировать на геном человека сотни миллионов фрагментов).
Этот подход очень удобен для повторного секвенирования геномов, которое проводится для выявления степени внутривидовых различий ДНК, анализа состава транскриптома (РНК-продуктов «считывания» генов) и выявления различия в нем на разных стадиях развития организма. Один из наиболее известных проектов в этой области – международный проект «1000 геномов», направленный на изучение редких и распространенных генных вариаций (полиморфизмов) в 14 популяциях человека на основе повторного секвенирования геномов свыше тысячи человек.
Проводим опознание
В последние годы было обнаружено, что вопреки первоначальным ожиданиям в геномах высших организмов доля ДНК, кодирующей белки, очень невелика. Для человека она составляет около 1,5 % или не более 30 тыс. генов. Структура нуклеотидных последовательностей этих генов прерывистая и содержит кодирующие (экзоны) и некодирующие (интроны) участки, а также регуляторные участки, с которыми связываются белки, запускающие процесс транскрипции (считывания ДНК).
Идентификация структуры гена – одна из наиболее актуальных задач биоинформатики, для решения которой используются методы машинного обучения (нейронные сети и другие подобные алгоритмы). В этом случае для известных достоверных последовательностей и структур генов предварительно рассчитываются наборы статистических параметров (частоты встречаемости определенных нуклеотидных фрагментов, корреляции между их расположением в последовательности, наличие регуляторных последовательностей и пр.), на основе которых и «обучают» программы распознавания генов.
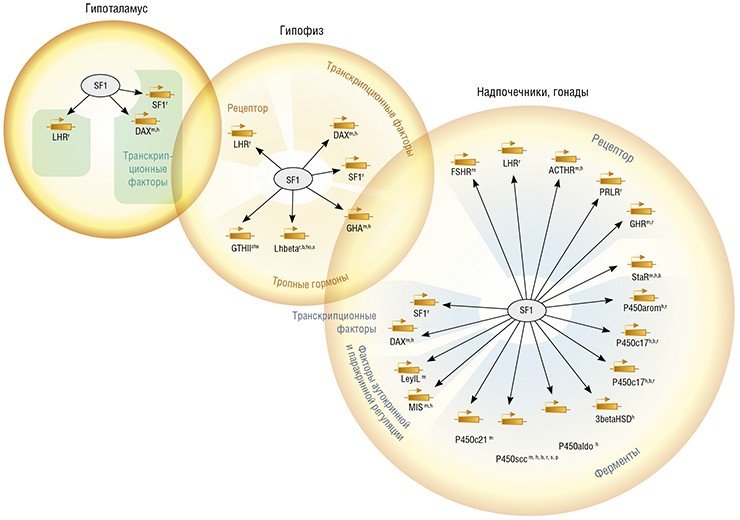
Однако наиболее ценную информацию для «опознания» генов дает сравнение нуклеотидной последовательности генома с последовательностями уже известных генов родственных видов. Такой же принцип широко используется и для предсказания функции «нового» гена: на основе гомологии (общности происхождения) ему приписывается известная функция родственного гена.
На сегодня имеется большое число баз данных, в которых дана функциональная аннотация генов или кодируемых ими белков. Например, база данных UniProt содержит наиболее полную информацию о белковых последовательностях и включает в себя свыше 500 млн объектов (для 14 % белков их функция была определена экспериментально, для 12 % – на основании данных по кодирующим их мРНК, для 70 % – предсказана по гомологии). Есть базы данных, в которых белки группируются по степени функциональной близости, например, база данных Pfam, содержащая свыше 14 тыс. белковых семейств, в которых объединены белки, выполняющие сходные функции.
Интенсивно развиваются и методы поиска сходных последовательностей в огромных массивах биологических баз данных, которые позволяют эффективно использовать для предсказания функции и структуры генов информацию по структуре и функции уже аннотированных генов и белков. Например, программа BLAST проводит поиск сходных последовательностей ДНК в банке данных нуклеотидных последовательностей общим размером 1,37×1012 символов всего за несколько минут.
Форма определяет функцию
Структурная биоинформатика изучает, соответственно, структурные свойства биологических макромолекул. Среди ее задач – сравнение и анализ структур белков и ДНК, распознавание функциональных участков молекул, компьютерное предсказание белковых взаимодействий.
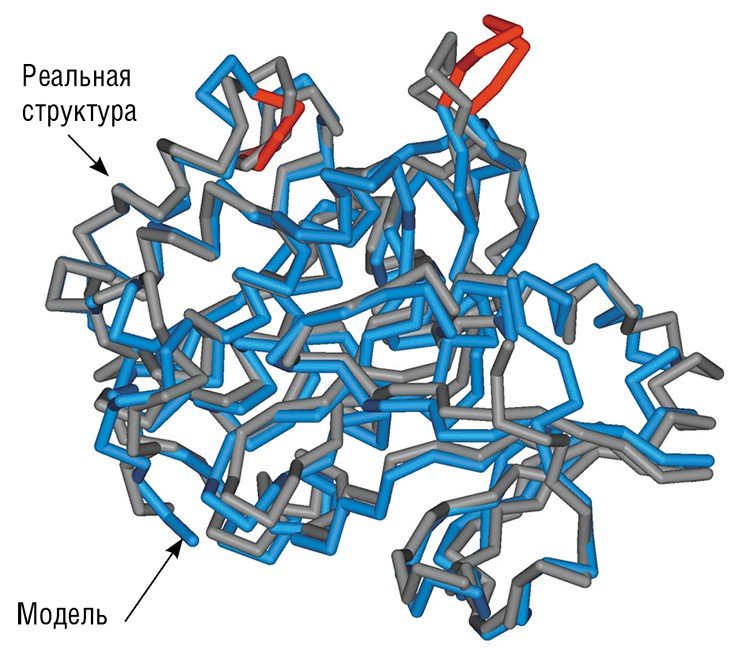
Пространственная структура белка, которая формируется в физиологических условиях в результате самостоятельной укладки полипептидных цепей, определяет и его функциональные свойства: наличие участков связывания малых химических соединений, ДНК, РНК и других белков. Информация о таких структурах хранится в банке данных Protein Data Bank, который уже сейчас содержит почти 90 тыс. моделей биологических макромолекул, включая не только сами белки, но и ДНК, РНК, а также их комплексы.
В этой связи для биологов очень важной является задача сравнения и классификации белковых структур. Методы структурной биоинформатики позволили разработать эффективные алгоритмы для парного и множественного сравнения белковых структур, а также создать свою белковую «систематику», т. е. классификацию на типы укладки полимерной цепи, структурные классы, семейства и подсемейства.
Такая классификация во многом способствует изучению эволюции белков и более глубокому пониманию их функций. Например, установлено, что в процессе эволюции изменения в пространственной структуре белков накапливаются гораздо медленнее, чем изменения в самих аминокислотных последовательностях. Кроме того, была сформулирована гипотеза о конечности числа возможных пространственных укладок полипептидной цепи белков – оно было оценено приблизительно в одну тысячу. Это предположение подтверждается исследованиями последних лет: число «опознанных» белковых структур растет ежегодно на 5—7 тыс., тогда как число белков с уникальной, ранее не известной структурой – всего на 100—150 штук.
Наиболее надежный способ получения моделей пространственных структур белков – рентгеновская кристаллография, однако он длительный, трудоемкий и дорогостоящий. Поэтому важным направлением структурной биоинформатики является разработка методов предсказания структуры белка по его аминокислотной последовательности. Для этого здесь, как и в компьютерной геномике, используются методы машинного обучения, а также технологии реконструкции пространственных структур «по гомологии», т. е. на основании сходства. В настоящее время наиболее точные предсказания структуры белка можно получить, если находится родственный ему белок с уже известной пространственной структурой. И чем выше будет степень родства двух белков, тем выше окажется точность модели.
Еще одна интересная область структурной биоинформатики – молекулярное моделирование структур биологических макромолекул. Современные алгоритмы, использующие технологии параллельных вычислений на высокопроизводительных компьютерных кластерах, позволяют рассчитывать системы, состоящие из десятков тысяч атомов! Это дает возможность в мельчайших деталях – на уровне отдельных атомов, исследовать эффекты влияния мутаций на структуру белка и характер взаимодействия его активных центров с метаболитами.
В генной «паутине»
Нужно отметить, что к концу XX в. в биологии сформировался интегральный взгляд на процессы в живых системах, которые рассматривались как результат совместного функционирования огромного количества клеточных компонентов, от низкомолекулярных метаболитов и макромолекул до клеточных структур.
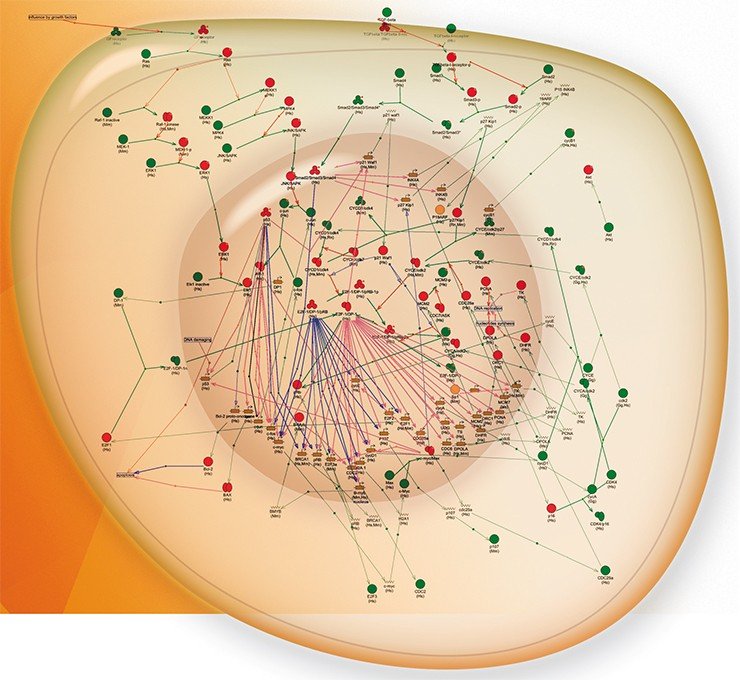
В этом ключе взаимодействия между компонентами живых клеток принято описывать в виде графов, узлами которых являются биологическое компоненты (гены, молекулы), а ребрами – взаимодействия между ними. Такие графы, именуемые генными сетями, описывают координированно функционирующие группы генов, которые контролируют развитие всех фенотипических признаков организма (Колчанов и др., 2008).
Такое представление межгенных взаимодействий – удобная математическая модель: на основе анализа структуры графа можно получать информацию о различных особенностях функционирования живых систем. В структуре графа можно выделить ряд важных элементов, в частности, положительные и отрицательные обратные связи, циклы, каскады передачи сигналов и т. д.
В случае, когда параметры взаимодействий между компонентами генной сети известны (например, оценены экспериментально), компьютерные программы позволяют построить кинетические модели, которые можно использовать для моделирования динамического поведения генных сетей, т. е. изменения концентрации компонентов этой сети в течение времени. Такие модели, уже позволившие получить ряд новых интересных данных, касающихся влияния мутаций на функции живых систем (Колчанов и др., 2008), имеют большое прикладное значение в биологии и медицине.
В свете эволюции
Сорок лет назад Ф. Добржанский (1973), один из основателей современной теории эволюции, отметил, что «в биологии ничто не имеет смысла кроме как в свете эволюции». Именно поэтому одна из основных областей применения информационных технологий в биологии – изучение молекулярной эволюции, которое заключается в построении моделей эволюции генов, учитывающих самые разные факторы: особенности структурной организации генов, пространственную структуру белков, взаимодействия белков с метаболитами, другими белками и ДНК, особенности функционирования генных сетей. Такие модели позволяют реконструировать эволюционную историю генов и белков, а на их основе эволюцию видов.
Современные модели накопления мутаций в геномных последовательностях используются для датировки эволюционных событий. Кроме того, модели эволюции позволяют оценивать влияние нуклеотидных и аминокислотных замен на структуру и функцию генов и кодируемых ими белков; это, в свою очередь, помогает оценивать влияние полиморфизмов, связанных с наследственными заболеваниями.
Характер накопления мутаций в генах свидетельствует об их функциональной важности: более важные гены, как правило, накапливают мутации с меньшей частотой, чем менее важные.
В лаборатории эволюционной биоинформатики и теоретической генетики Института цитологии и генетики СО РАН (Новосибирск) проведен анализ эволюции генов, вовлеченных в функционирование клеточного цикла – одного из ключевых процессов, обеспечивающих рост и деление клеток. Контроль за этим процессом осуществляется семейством специфических белков – циклинов, которые в свою очередь вовлечены в целую сеть взаимодействий с другими генами.
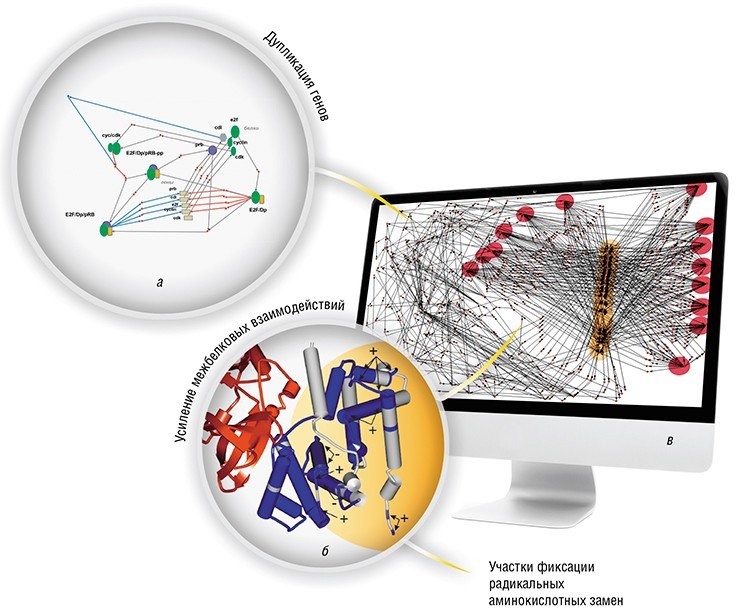
На основе реконструкции и сравнения генных сетей контроля клеточного цикла млекопитающих и грибов удалось выявить молекулярно-генетические механизмы эволюционного усложнения этой генной сети в процессе эволюции.
Во-первых, это массовые дупликации генов, существенно увеличивающих число белков (циклинов и взаимодействующих с ними циклин-зависимых киназ), функционирующих в генной сети. Во-вторых, на поверхностных участках циклинов происходит накопление радикальных аминокислотных замен на стороне, противоположной месту их контакта с циклин-закисимыми киназами. На основе всех этих изменений происходит увеличение интенсивности белок-белковых взаимодействий и, как следствие, усложнение генной сети за счет существенного роста числа регуляторных петель с обратными связями (Gunbin et al., 2010; Турнаев и др., 2009).
Экстрактор информации
Бурное развитие экспериментальных методов исследований в биологии, биомедицине и биотехнологии сопровождалось резким скачком в объеме получаемых новых знаний и, как следствие, научных публикаций. В настоящее время в базе данных PubMed – официальном хранилище публикаций биологического и биомедицинского профиля – содержится более 20 млн рефератов научных статей. Число публикаций растет столь быстро, что всю имеющуюся на сегодня информацию принципиально невозможно проанализировать без использования компьютерных средств. Поэтому в мире активно развиваются методы интеллектуального анализа данных, направленные на извлечение информации из научных текстов.
Такой компьютерный анализ текстов часто называют текст-майнинг (от англ. text mining, «добыча» знаний из текстов). В этих технологиях широкое применение нашли методы семантических правил или шаблонов. В веб-программировании семантический шаблон представляет собой регулярное выражение (формальное описание задачи поиска в тексте данных, отвечающих определенным условиям), где порядок встречаемости различных концептов отражает последовательность слов в предложении, на основании которого можно сделать вывод о наличии факта взаимодействия двух или более объектов, описанных в этом предложении.
В лаборатории компьютерной протеомики ИЦиГ СО РАН разработана первая в России компьютерная система автоматической экстракции знаний из текстов научных публикаций (Demenkov et al.; 2012 Деменков и др., 2008). ANDSystem состоит из модуля компьютерного анализа текстов, базы знаний ANDCell, а также программы визуализации результатов в виде ассоциативных семантических сетей ANDVisio. Вершинами таких сетей являются молекулярно-генетические объекты, заболевания и процессы, а связями между ними – типы взаимодействий и ассоциаций.
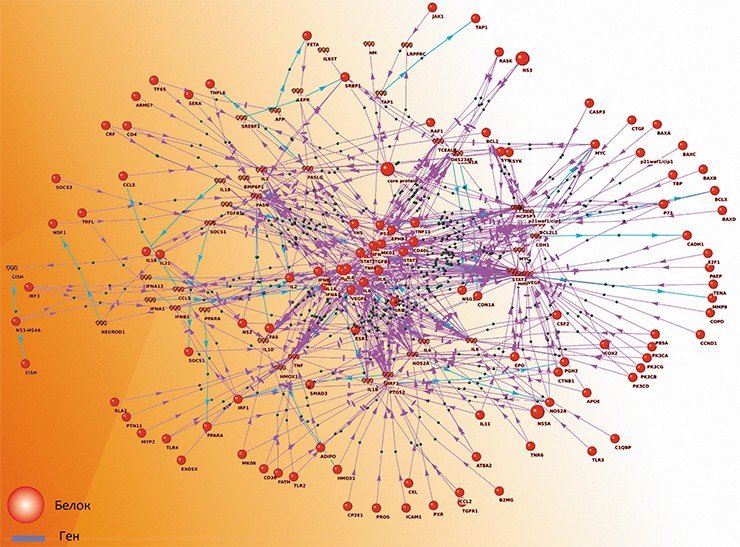
Было создано более 2 тыс. семантических шаблонов, использующих специально разработанную онтологию молекулярно-генетических взаимодействий и генетической регуляции, содержащую ряд словарей, включающих названия макромолекул, метаболитов, биологических процессов и заболеваний, а также различных типов взаимоотношений между ними. Система обладает дружественным интерфейсом пользователя со многими функциями, включая отсылку на сайты молекулярно-генетических баз данных, а также рефераты статей, из которых была экстрагирована информация.
Применение текст-майнинга к анализу публикаций из базы данных PubMed позволило получить информацию относительно более чем 5 млн фактов, касающихся молекулярно-генетических событий в клетках различных тканей и организмов. Эти знания имеют чрезвычайно большое значение для автоматизации процесса реконструкции генных сетей.
Система ANDSystem также активно используется для интерпретации экспериментальных данных. Например, была проведена реконструкция и анализ сетей молекулярно-генетических взаимодействий ряда белков у различных штаммов бактерии Helicobacter pylori, выделенных у пациентов с хроническими гастритами и опухолями желудка. Показано, что различия в экспрессии этих белков могут быть связаны с адаптацией бактерий к различным условиям среды, т. е. человеческого желудка (Momynaliev et al., 2010).

С помощью ANDSystem были обнаружены кластеры белков, которые могут участвовать в процессах адаптации организма человека к экстремальным условиям, в том числе к условиям невесомости (Ларина и др., 2012; Пастушкова и др., 2012); описаны молекулярные механизмы взаимосвязи между миопией и глаукомой, предложены новые молекулярные маркеры этих заболеваний (Подколодная и др., 2010) и т. п.
В настоящее время с использованием ANDSystem ведутся работы по реконструкции и анализу молекулярно-генетических сетей, вовлеченных в жизненный цикл вируса гепатита С в рамках европейского международного проекта FP7.
Биоинформатику, возникшую на стыке информационных технологий и биологии, поначалу рассматривали как средство поддержки научных исследований. Однако со временем становилось все более очевидным, что эта наука – важная и неотъемлемая часть биологии, без которой ее дальнейшее развитие просто невозможно себе представить.
Тесный союз биологии и информационных технологий обеспечивает одновременный бурный рост обеим этим научным дисциплинам. Необходимость решать новые широкомасштабные биологические задачи требует создания все более производительных алгоритмов для анализа данных и увеличения вычислительных мощностей компьютеров. Это, в свою очередь, дает возможность ставить новые эксперименты и получать новые знания, углубляющие наши представления о структуре и функционировании биологических объектов.
Литература
Деменков П. С., Аман Е. Э., Иванисенко В. А. Associative Network Discovery (AND) – компьютерная система для автоматической реконструкции сетей ассоциативных знаний о молекулярно-генетических взаимодействиях // Вычислительные технологии. 2008. Т. 13, № 2. С. 15—19.
Ларина И. М., Колчанов Н. А., Доброхотов И. В. и др. Реконструкция ассоциативных белковых сетей, связанных с процессами регуляции обмена и депонирования натрия в организме здорового человека на основе изучения протеома мочи // Физиология человека. 2012. Т. 38, №3. С.107—115.
Подколодная О. А., Яркова Е. Э., Деменков П. С. и др. Использование компьютерной системы ANDCell для реконструкции и анализа ассоциативных сетей потенциальных механизмов взаимосвязи миопии и глаукомы // Информационный вестник ВОГиС. 2010. Т. 14, № 1. С. 106—115.
Системная компьютерная биология / Под ред. Колчанова Н. А., Гончарова С. С., Лихошвая В. А., Иванисенко В. А., Новисибирск: Изд-во СО РАН, 2008.
Momynaliev K. T., Kashin S. V., Chelysheva V. V. et al. Functional Divergence of Helicobacter pylori Related to Early Gastric Cancer // J. Proteome Res. 2010 Jan; 9(1):254—67.
Gunbin K. V., Suslov V. V., Turnaev I. I. et al. Molecular evolution of cyclin proteins in animals and fungi // BMC Evol. Biol. 2011. V. 11. P. 224.
Материал подготовлен при поддержке проекта «Научные школы» НШ-5278.2012.4