Как читают гены
Истоки современных методов чтения наследственной информации лежат в конце 60-х – начале 70-х гг. прошлого века. Сама история создания гигантских автоматизированных «фабрик по чтению геномов» весьма поучительна: здесь сталкивались «романтики», готовые неопределенное время искать совершенного «журавля в небе», и прагматики, требовавшие прогресса – пусть небольшого, но в обозримом будущем. В результате колоссальной работы ученых, удалось на порядки снизить стоимость и ускорить процесс дешифровки ДНК
Научные методы, методики, технологии... Зачем их вообще нужно рассматривать, да еще далекому от науки человеку? Но «бытие определяет сознание», как верно говорил классик марксизма-ленинизма, а бытие ученого – это, прежде всего, инструменты и методы, которыми он пользуется. Они же в каждый конкретный момент определяют пределы возможного и доступного...
Истоки современных методов чтения наследственной информации лежат в конце 60-х – начале 70-х гг. прошлого века. И путь был тернист: здесь сталкивались «романтики», готовые неопределенное время искать совершенного «журавля в небе», и прагматики, требовавшие прогресса – пусть небольшого, но в обозримом будущем. И может быть, точка в этом споре поставлена лишь временно...
На заре генетики гены – материальные носители наследственности, определяющие признаки и особенности организма, – были такой же абстракцией, как и эфир в физике. Возможность «читать гены», т.е. определять нуклеотидные последовательности ДНК и РНК, изменила генетику до неузнаваемости, фактически создав новую науку – геномику. О революционном значении этого события свидетельствует хотя бы присуждение в 1980 г. Нобелевской премии по химии английским исследователям У. Гилберту и Ф. Сэнгеру, разработавшим в 1970-е гг. методы определения нуклеотидных последовательностей, названные в честь их авторов и широко используемые до сих пор.
Начало нового века ознаменовалось очередным витком в развитии геномных исследований. Что ни день, то расшифровываются геномы все новых и новых организмов, причем найти неизученные объекты становится непросто: исследователи в шутку сетуют, что скоро секвенировать (так называют определение нуклеотидных последовательностей люди, которые этим профессионально занимаются) будет нечего. Одновременно значительно расширились сферы применения секвенирования. Теперь это инструмент для самых разнообразных исследований: от поиска маркеров наследственных заболеваний до оценки разнообразия организмов, населяющих те или иные экосистемы.
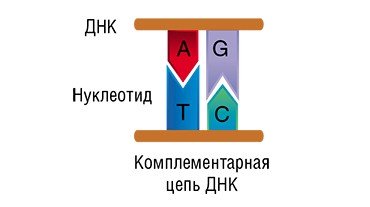 Все современные технологии «чтения» ДНК основаны на ее удивительном свойстве – комплементарности.
Все современные технологии «чтения» ДНК основаны на ее удивительном свойстве – комплементарности.
Одиночная нить ДНК представляет собой природный полимер – «бусы», состоящие из 4-х видов бусинок-нуклеотидов: аденина (A), тимина (T), гуанина (G) и цитозина (C). Нуклеотиды и являются «буквами» генетического кода: каждый триплет кодирует одну аминокислоту; а последовательность нуклеотидов, называемая геном, – один белок.
Две нити ДНК удерживаются вместе за счет взаимодействий между неслучайными парами нуклеотидов, подходящих друг другу, как ключ к замку: A – к T, G – к C.
Благодаря этому на нити ДНК может быть синтезирована дочерняя комплементарная цепь. Таким способом происходит как копирование генетической информации, так и считывание ее для синтеза белка
Такие успехи в расшифровке геномов были достигнуты благодаря, в первую очередь, экстенсивному развитию традиционных методов секвенирования ДНК и созданию многочисленных центров «чтения геномов». Располагая многочисленным квалифицированным персоналом, они в каком-то смысле являются аналогами мануфактур, где автоматизация затронула лишь последние стадии «производственного процесса».
И хотя основные результаты «чтения геномов» были получены и до сих пор получаются именно в таких центрах, ученые всегда стремились к более полной, комплексной автоматизации, а в перспективе – даже к полностью роботизированному «производству».
А теперь более подробно остановимся на всех этапах эволюции методов «чтения» наследственности.
Геном на вес золота
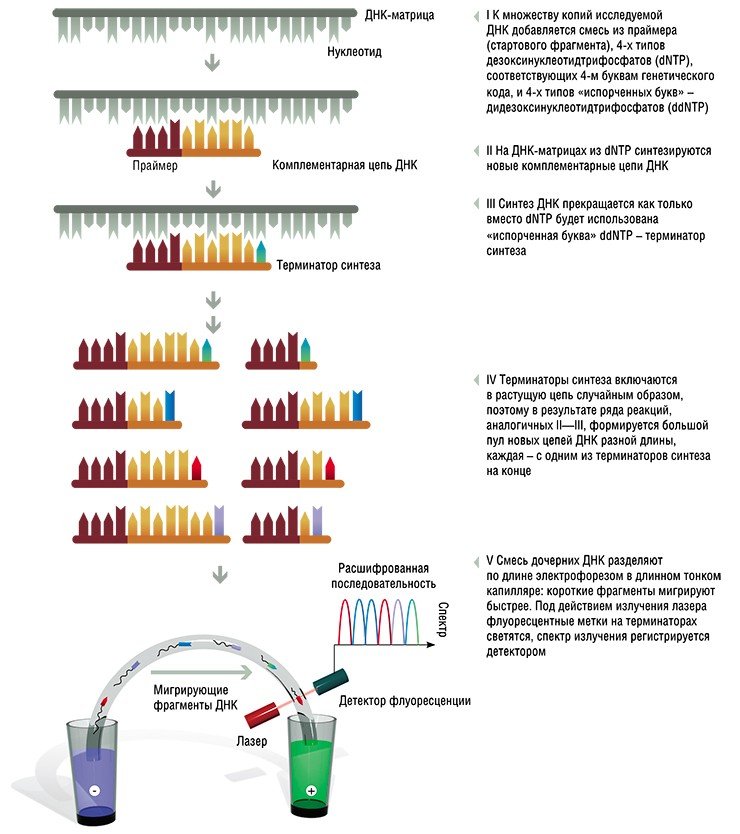
Так называемая реакция Сэнгера, лежащая в основе ставшего классическим метода «чтения ДНК», заключается в том, что на исследуемом фрагменте ДНК (матрице) синтезируется ряд дочерних цепей разной длины. Все они оканчиваются особым нуклеотидом-терминатором, комплементарным одной из «букв» матрицы. При этом терминаторы помечены разными флуоресцентными метками, благодаря чему возможна их детекция.
Здесь следует заметить, что большинство объектов, с которыми имеют дело генетики, представляют собой весьма длинные нуклеотидные последовательности. Геномы бактерий, даже самых простых, редко бывают короче миллиона пар оснований. Геном человека – более 3 млрд пар оснований, и даже относительно небольшая нуклеотидная последовательность генома человеческой митохондрии (клеточной «электростанции», имеющей собственную наследственную информацию) превышает в длину 16 тыс. пар оснований.

Главным недостатком методов Сэнгера и Гилберта является жесткое ограничение на длину последовательности, определяемой в одном эксперименте, – не более 1000 пар оснований. Причем это ограничение вытекает из принципов метода, и в этом смысле оно неустранимо. Чтобы «читать» реальные геномы с помощью такого метода, необходимо сначала направленно «расчленить» исходные объекты на перекрывающиеся мелкие фрагменты, а затем из их последовательностей реконструировать исходную.
Для генома человека, например, число таких фрагментов должно составлять десятки миллионов! Подобное ограничение значительно осложняет расшифровку реальных геномов, а также создает трудности при попытках автоматизации этого процесса.
Нужно отметить, что сам анализ продуктов реакции Сэнгера легко автоматизируется за счет использования флуоресцентных меток и капиллярного электрофореза. Автоматизация позволила значительно снизить затраты труда на эту стадию. Однако длина определяемой в одном эксперименте последовательности при этом несколько уменьшилась: на заре становления методов эксперты читали до 1200—1500 «букв», современные автоматы – не более 700—900. Но дело даже не в том: основной труд приходится как раз на «расчленение» исходной последовательности и последующую ее сборку.
Масштабы необходимых для этих операций людских и материальных ресурсов позволили в свое время сравнивать по затратам проект «Геном человека» с проектом высадки на Луну. Стоимость расшифровки первого чернового варианта генома человека, законченной в 2001 г., оценивалась в 300 млн долл., а его окончательный вариант вместе с разработкой необходимых технологий обошелся примерно на порядок дороже. Правда, черновой вариант третьего генома приматов (макаки-резуса), опубликованный в феврале 2007 г., стоил «всего» 22 млн долл.
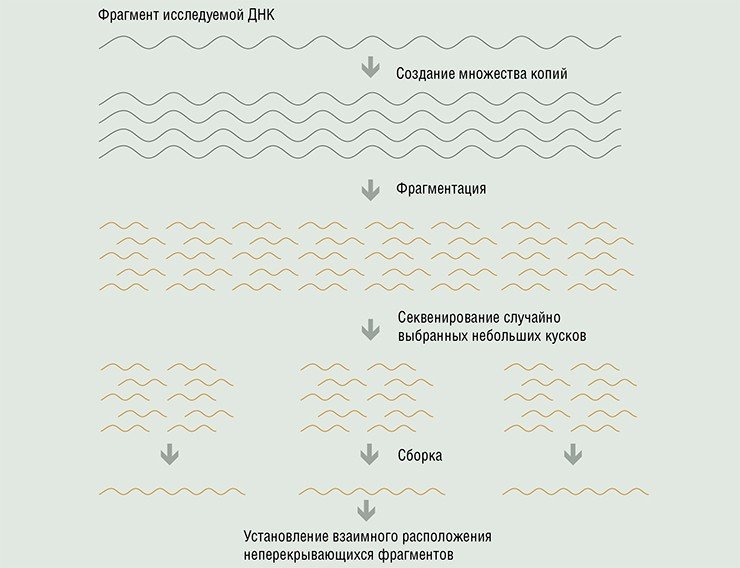
Помимо огромной трудоемкости, процесс расчленения-сборки имеет еще один существенный недостаток – проблему аутентичности получаемых результатов. Сложно гарантировать, что все десятки миллионов фрагментов, получаемых многократным и многоэтапным копированием участков исходной ДНК, остаются ее точной копией.
Кстати сказать, первоначально опубликованная последовательность генома человека была «мозаичной», т. е. представляла собой своего рода «лоскутное одеяло» из фрагментов геномов разных индивидуумов (по всей видимости, граждан разных стран и даже представителей разных рас). Правда, полагают, что основной вклад в эту мозаику внес геном Крейга Вентера, президента корпорации Celera Genomics.
Журавль или синица?
Все недостатки методов секвенирования ДНК стали очевидны сразу же после начала их практического применения. Поэтому правительство США направило большие силы и средства (даже из бюджета оборонного ведомства) на разработку альтернативных подходов, которые были бы свободны от ограничений на длину анализируемых фрагментов. Большинство таких подходов предполагало работу с одной молекулой ДНК, а не с набором ее копий-потомков, что одновременно снимало проблему аутентичности.
Первоначально наиболее известные проекты секвенирования единичных молекул ДНК без принципиальных ограничений на их длину предполагали использование двух подходов: сканирующую туннельную микроскопию либо последовательное отщепление нуклеотидных остатков от молекулы ДНК с помощью специального фермента эндонуклеазы.
С помощью туннельного микроскопа исследователи намеревались «заглянуть» в большую бороздку распластанной на подложке ДНК и «прочесть» последовательность, используя разницу в геометрии отдельных «букв»-оснований. К сожалению, разрешающей способности этого метода оказалось недостаточно, хотя сегодня он широко используется, например, для детального исследования комплексов ДНК с различными (в том числе и совсем небольшими) молекулами.
Намного более обещающим казалось использование экзонуклеазы для медленного пошагового гидролиза (расщепления) единичной молекулы ДНК. Исследователям довольно легко удалось научиться помещать в реактор одну молекулу ДНК и контролировать скорость реакции. Но вот разработать способ достоверной идентификации полученных единичных нуклеотидов, к сожалению, так и не удалось.
Однако практика настоятельно требовала дальнейшего удешевления секвенирования с учетом уже отработанной полной автоматизации самой реакции Сэнгера. И устав ждать «журавля в небе» от создателей принципиально новых подходов, независимые группы исследователей и ведущие фирмы-разработчики взялись за автоматизацию этапа трудоемкой подготовки к классическому секвенированию, а именно – автоматизацию процесса расчленения одной длинной молекулы ДНК на много мелких фрагментов заданной длины.
Поскольку при таком подходе фрагменты нарезаются случайным образом, то для достоверного чтения длинных последовательностей их требуется значительно больше, чем при направленном фрагментировании ДНК. Поэтому требовалось автоматизировать разделение этих фрагментов в пространстве и последующую регистрацию нескольких миллионов реакций секвенирования, происходящих одновременно. Так появилось новое поколение приборов для определения нуклеотидных последовательностей, получившее название «системы массового параллельного секвенирования».
Разделяй и секвенируй
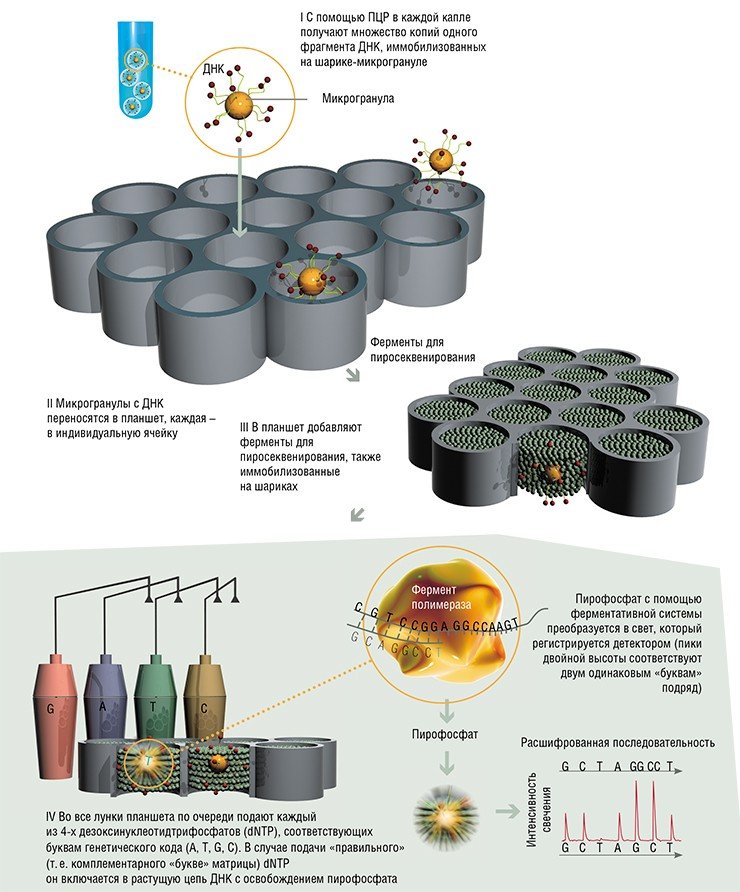
Группа разработчиков, финансируемая Национальными институтами здравоохранения США (проект Life Science 454), предложила для разделения фрагментов использовать микрогранулы, взвешенные в отдельных каплях водно-масляной эмульсии. При этом микрогранулы должны быть изолированы друг от друга и содержать не более одной молекулы фрагмента исходной ДНК. После амплификации, т.е. создания внутри гранулы «колонии» из огромного числа точных копий исходного фрагмента с помощью полимеразной цепной реакции (ПЦР*), можно приступать к самому секвенированию.
На этом этапе гранулы помещают в специальный планшет, в каждой ячейке которого есть место только для одной гранулы. Для визуализации каждого шага реакции секвенирования используется давно известное пиросеквенирование – метод, в котором при подаче в ячейку «правильного» нуклеотидтрифосфата, который включается в растущую цепь ДНК, испускается свет. Изображение планшета постоянно записывается, так что на каждой стадии можно регистрировать свет, испускаемый отдельными ячейками.
После того как прототип такого прибора доказал свою работоспособность, разработку купил концерн Roche, который и занимается коммерческим распространением прибора.
Другой подход к решению проблемы разделения в пространстве «молекулярных колоний» предложен в рамках проекта SOLEXA. В этом случае раствор фрагментов исходной ДНК наносится на подложку, к которой прочными ковалентными связями присоединены олигонуклеотиды – «затравки» для последуюшего проведения ПЦР. Раствор ДНК разбавляется настолько, чтобы между отдельными молекулами имелось достаточное расстояние. Копии одного фрагмента (молекулярная колония) остаются в той же области, где находился исходный фрагмент, поскольку при их синтезе используются находящиеся рядом на поверхности подложки олигонуклеотиды.
После демонстрации практических успехов этого подхода разработку приобрела и успешно распространяет биотехнологическая компания Illumina. Аналогичная разработка SOLiD, находящаяся сейчас на стадии коммерческого внедрения, имеется и у монополиста на рынке традиционного секвенирования – фирмы ABI.
В результате создания подобных технологий расшифровка нуклеотидной последовательности единичного фрагмента исходной ДНК теперь стоит совсем дешево и не требует ручного труда. Правда, пока длина расшифрованных фрагментов невелика – около 400 нуклеотидных остатков для Life Science 454, и лишь несколько десятков – для SOLEXA и SOLiD. Но зато таких фрагментов может быть очень много…
К сожалению, первые итоги практического использования этих приборов показали, что надежды, которые с ними связывали, оправдались далеко не в полной мере. Некоторые успехи были продемонстрированы лишь при дешифровке относительно простых геномов бактерий. Что касается крупных геномов высших организмов, включая человека, то некоторые их участки совсем или почти не представлены среди расшифрованных последовательностей. При этом небольшая длина фрагментов делает сборку из них протяженных последовательностей задачей крайне сложной и принципиально неоднозначной.
Тем не менее эти технологии позволили практически в неограниченных масштабах проводить повторное секвенирование и сравнительный анализ геномов разных особей с тем, чтобы, к примеру, выявить индивидуальные различия, что очень важно для медицинских целей. Чтение же больших геномов de novo (т.е. «с чистого листа») пока по-прежнему не может обойтись без работы огромных «фабрик по секвенированию».
Геном задаром?
Научные лаборатории и биотехнологические компании продолжают активно соревноваться друг с другом в стремлении первыми предоставить «дешевые геномы», и ставки в этом соревновании весьма высоки.
Так, в сентябре 2003 г. научный фонд К. Вентера посулил полмиллиона долларов тому, кто первым прочтет геном человека за одну тысячу долларов. В 2004 г. национальные институты здравоохранения США запустили программу, предусматривающую 70 грантов (каждый – по миллиону долларов) для поддержки исследований по снижению стоимости расшифровки больших геномов до 100 тыс. долл. и менее. А еще через два года фонд Вентера и X Prize Foundation объявили о премии в 10 млн долл. исследователям, которые сумеют расшифровать 100 человеческих геномов за 10 дней по цене не более 10 тыс. долл. за геном.
Пока обещанные призовые не получил никто: на практике расшифровка индивидуальных геномов обходится намного дороже. Так, год назад на торжественной церемонии в Хьюстоне Джеймсу Уотсону, одному из легендарных открывателей структуры ДНК, были вручены два DVD с записью его генома. По признанию дарителей, эта работа хотя и была сделана относительно быстро – всего за два месяца, но оказалась весьма затратной: стоимость одних лишь использованных реактивов составила около миллиона долларов!
Что же означают рекламные призывы на сайтах некоторых биотехнологических компаний – «Секвенируем ваш геном! Недорого!»? Может быть, они знаменуют собой начало новой эры – эры секвенирования индивидуальных геномов, перехода генетики с уровня видов и популяций на уровень индивидуумов? Смогут ли ученые достичь заданной ценовой планки за расшифровку полного человеческого генома, и если да, то с помощью каких методов?
Скорее всего, это будут все же не столь популярные сегодня приборы массового параллельного секвенирования. Эволюция снова сделает виток, и очередными «generation next» станут устройства, работающие с единичными молекулами нуклеиновых кислот. Но это уже тема для будущей статьи.
Литература
Bentley D.R. et al. Accurate whole human genome sequencing using reversible terminator chemistry. // Nature. – 2008. – V. 456(6). – P. 53–59.
Wang J. et al. The diploid genome sequence of an Asian individual. // Nature. – 2008. – V. 456(6). – P. 60–66.
Фото из архива ЦКП «Секвенирование ДНК» СО РАН (Новосибирск)
* ПЦР – метод, позволяющий производить в неограниченных количествах копии одной молекулы нуклеиновой кислоты с помощью специальных ферментов. Метод базируется на способности нуклеиновых кислот к комплементарным взаимодействиям